利用機器學習在基因組層面預測食源性沙門氏菌的耐藥性
發布時間:2024-09-14 瀏覽次數:619
AMR是一種全球性的健康危機,給食品生產和安全、醫療保健、公共衛生和國家經濟都帶來了多方面的挑戰。盡管AMR本質上是一種自然進化的過程,但是抗生素的使用是耐藥菌的出現和傳播的一大驅動力。另外,農業環境中抗生素的濫用或者誤用是造成沙門氏菌耐藥的主要驅動力。隨著測序技術的進步,全基因組測序(WGS)已成為常規流行病學監測的一部分。在數據分析方面,機器學習已成為分析測序平臺產生的大型數據集的首選方法。然而,當前卻鮮有研究將基于機器學習的數據分析技術應用在食源性致病菌的耐藥性預測上。基于此,Edmund O. Benefo等人開發一種基于基因組的機器學習模型,用于預測來自雞肉的沙門氏菌的AMR,重點關注了八種抗菌劑,包括阿莫西林-克拉維酸、氨芐西林、頭孢菌素、頭孢曲松、磺胺異惡唑、鏈霉素、四環素和頭孢西丁。
研究共收集了雞源沙門氏菌405株,取205株菌的表型和基因組信息用于機器學習模型的搭建,剩下200株作為機器模型的性能表征。研究訓練了四種機器學習算法:集成分類器、隨機森林、支持向量機和極端梯度提升去分析菌株中的基因存在或缺失,并將其與表型關聯。在模型構建過程中,將菌株進一步按照7:3規劃訓練集和驗證集,利用靈敏度、特異性、數據平衡準確性及科恩的κ統計值,針對每一抗生素篩選出表現性能最好的模型(表1)。然后,作者分別利用這些構建好的模型對剩余200株菌株的表型進行預測,并將其結果與ResFinder數據庫中的預測結果進行比較。結果顯示,模型預測的耐藥性流行率從1%(頭孢曲松)到65.5%(鏈霉素)不等。與ResFinder的預測相比,本研究的預測準確度超過95%,見圖1和表2。另外,作者還分別列舉出了針對每種抗生素進行預測AMR的耐藥決定因子,盡管有些決定因子現今還未得到驗證。
總的來說,文章證明了機器學習在預測食源性病原菌AMR的潛力,突出了機器學習方法在理解AMR機制和預測耐藥模式方面的適用性和可靠性。然而,文章也有幾點問題值得指出:(1)文章最后更多地關注于和ResFinder的比較,而實際上應該給出此預測方法與標準方法的比較結果;(2)研究列舉了機器算法給出的權重位于前10的耐藥決定因子,但是有些因子是否真的和耐藥表型相關,在研究中并未得到證明。因而,未來在基于機器學習算法的耐藥性預測方面,開展一些基因表達或者敲除實驗去驗證這些新的耐藥決定因子是非常必要的。
表1 針對8種抗生素的機器學習最佳模型及其預測性能展示
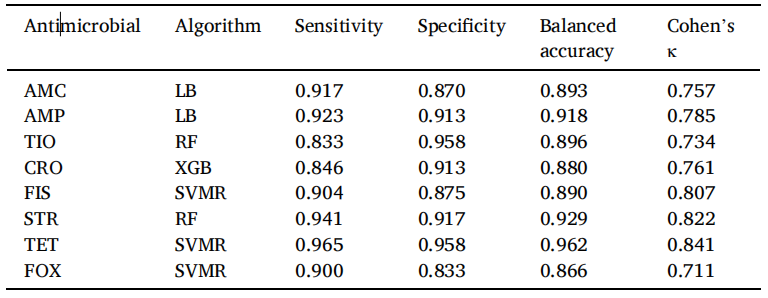
表2 基于本研究機器學習模型和基于ResFinder數據庫的預測性能比較
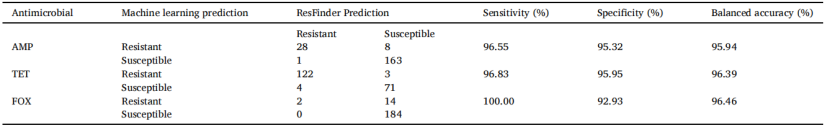
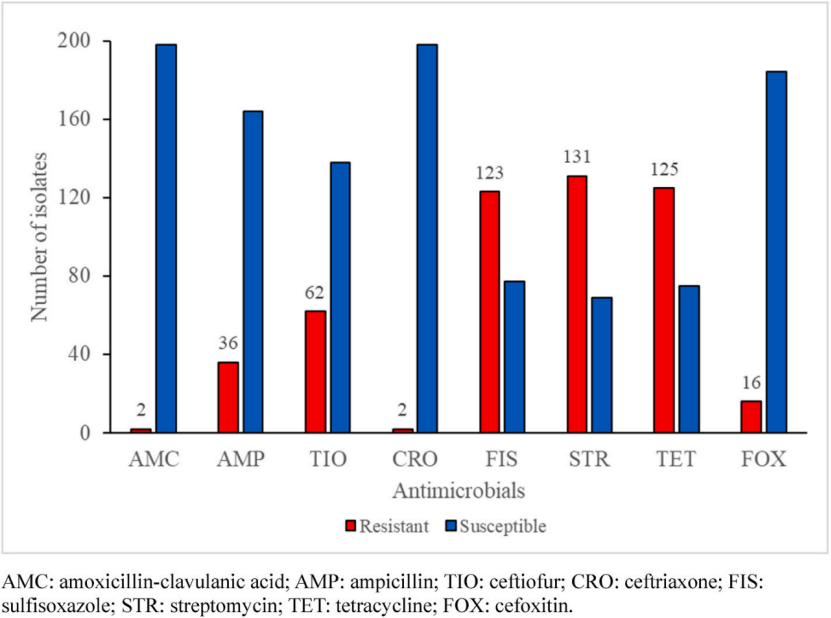
圖1 所建立的機器學習模型給出的200株沙門氏菌中AMR流行率統計結果
原文鏈接:https://doi.org/10.1016/j.lwt.2024.116122
來源:微生物安全與健康網,作者~鄒晶晶。






